- 영양 조건에 따른 미생물 성장 핵심 대사 반응 규명 -
미생물 성장의 핵심 대사반응(metabolic reaction: 세포 내 대사물질이 생성 및 분해되는 생화학 과정)을 규명할 수 있는 인공지능 기술이 개발됐다.
한국연구재단(이사장 이광복)은 윤성호 교수(건국대학교) 연구팀이 인공지능과 가상세포(생명체의 전체 대사과정을 컴퓨터상으로 구현함. 컴퓨터 모의실험을 통해 생명현상을 연구하거나 설계하는 기술 ) 기술을 활용하여 다양한 영양 조건에서 미생물 성장을 촉진하거나 저해하는 대사반응을 규명했다고 밝혔다.
미생물은 주어진 영양분을 효율적으로 이용하기 위해 세포 내 대사과정을 정밀하게 조정함으로써 최적의 세포 성장을 유지한다. 이러한 세포의 대사과정을 이해하기 위해서는 성장을 촉진하거나 저해하는 대사 유전자 및 경로를 식별하는 것이 중요하다.
그러나 수 천 개의 유전자, mRNA, 단백질, 대사물질들이 서로 복잡하게 얽혀 있는 미생물 시스템 내에서 세포 성장에 직접적으로 영향을 미치는 대사반응을 실험적으로 규명하는 것은 많은 시간과 자원을 필요로 한다.
연구팀은 가상세포로부터 예측된 대사반응 데이터와 다양한 성장 데이터를 통합 분석하는 머신러닝 및 딥러닝 모델을 개발, 연구에 널리 쓰이는 모델 미생물인 대장균 K-12를 대상으로 30가지 주요 영양조건에서의 균주 성장에 중요 또는 저해되는 대사경로를 도출했다.
그 결과, 생합성(생명체가 유기물질을 합성하는 대사과정) 경로는 대부분의 탄소원(생명체가 에너지를 얻고 성장하기 위해 필요한 탄소를 포함한 물질)에서 성장을 촉진하지만, 에너지 생성 경로의 중요도는 탄소원에 따라 달라진다는 것을 확인했다.
또한, 예측된 주요 대사반응은 유전자 조작 실험과 배양실험을 통해 검증했다. 예를 들어 대장균이 아세트산을 탄소원으로 이용할 경우, 피루브산 산화과정(세포의 주 에너지 생산 경로인 해당과정(glycolysis)과 TCA 회로(tricarboxylic acid cycle)를 연결해주는 대사반응)이 균체 성장을 저해한다고 예측되었으며, 실제로 이를 차단하면 성장이 촉진됐다.
이번 연구성과에 대해 윤성호 교수는 “유전체 설계를 통한 맞춤형 미생물 제작 및 최적의 생산 전략 수립에 중요하게 이용될 수 있다”며, “향후 다양한 생명현상 연구에 확대 적용될 수 있으리라 기대된다”고 밝혔다.
제 1저자인 우현재 석사과정생은 “가상세포 기반 예측 데이터와 대량의 실험데이터를 통합 분석하는데 인공지능 기법이 효율적으로 이용될 수 있음을 증명했다”고 덧붙였다.
과학기술정보통신부와 한국연구재단이 추진하는 기초연구사업 중견연구와 기본연구의 지원으로 수행된 이번 연구 성과는 시스템생물학 분야 국제학술지 ‘몰레큘러 시스템즈 바이올로지(Molecular Systems Biology)’에 1월 30일 온라인 게재되었다.
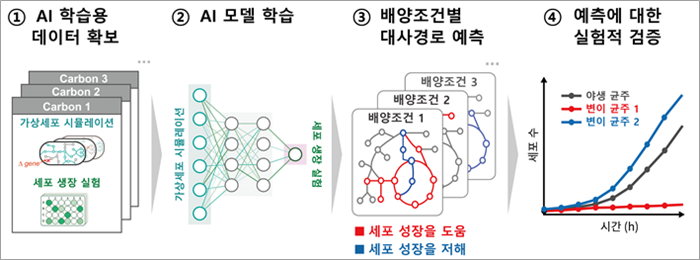
(그림1) 인공지능(AI)을 통한 핵심 대사반응 규명 과정
① 인공지능 모델을 훈련시키기 위해, 30가지 영양조건에서 각각 1,422개의 대장균 유전자를 제거했을 때의 성장값을 출력데이터로 사용하고, 이에 해당하는 대사경로 시뮬레이션 값을 입력데이터로 이용함. ② 두 가지 인공지능 모델(머신러닝, 딥러닝)을 생성함 ③ 각 30가지 영양조건에서의 미생물 성장에 중요/저해되는 대사경로를 인공지능 모델을 통해 예측함. ④ 주요 예측 대사반응에 대해 유전자 제거 및 배양실험을 통해 검증함. [그림설명 및 그림제공 : 건국대학교 윤성호 교수]
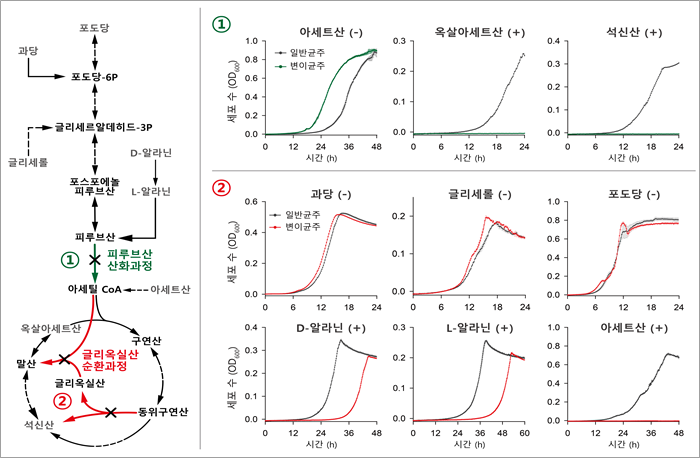
(그림2) 인공지능 모델로부터 예측된 주요 대사반응의 실험검증
특정 대사반응은 탄소원에 따라 세포 성장을 촉진(+) 또는 저해(-)한다고 예측되었으며, 이를 유전자 조작 및 배양실험을 통해 검증함. (좌측 그림): 유전자 제거로 의해 차단된 대사반응을 X로 표시함. (우측 그림): 각 대사반응이 제거된 균주와 야생형(wild-type) 균주의 성장을 비교함. ① 피루브산 산화과정(pyruvate dehydrogenase)은 옥살아세트산과 석신산에서 성장이 촉진(+)된다고 예측되었으며, 이 대사반응을 제거하면 성장이 저해됨. 반대로, 성장을 저해한다고 예측된 아세트산(-)에서는 피루브산 산화과정을 제거하면 성장이 촉진됨. ② 글리옥실산 회로(gloxylate shunt)를 차단하면, 알라닌(+)과 아세트산(+) 배양환경에서는 미생물 성장이 저해되고, 포도당(-), 과당(-), 글리세롤(-)에서는 성장이 촉진짐. [그림설명 및 그림제공: 건국대학교 윤성호 교수]